4 Datenstruktur
| Thema | Inhalte |
|---|---|
| Werte, Vektoren, Listen | chr, num, logi, c(), list(), mode(), coercion, Abruf von Elementen, list(list()) |
| Workspace | rm(), Besen |
| Berechnungen | Übersicht Berechnungsfunktionen, z-Standardisierung |
| Matrizen | matrix(), 2D Indizierung |
| tidy Daten | Zeilen: Beobachtungen, Spalten: Variablen |
tidyverse |
Installation und library(package) |
data.frame und tibble |
Unterschiede, as.data.frame(), as_tibble(), $, [], Zugriff auf Zeile, Spalte & Zelle, Zeilennamen |
| Daten laden und speichern | Import per klick, read./{*}, sep=, dec=, .xlsx, .svs, write./{*}.csv() |
| Daten anschauen | View(), head(), str(), count() |
4.1 RMarkdown
Das R Markdown Skript ist ein besonderes Dateiformat für R Skripte. Es enthält Fließtext und eingebetteten R Code:
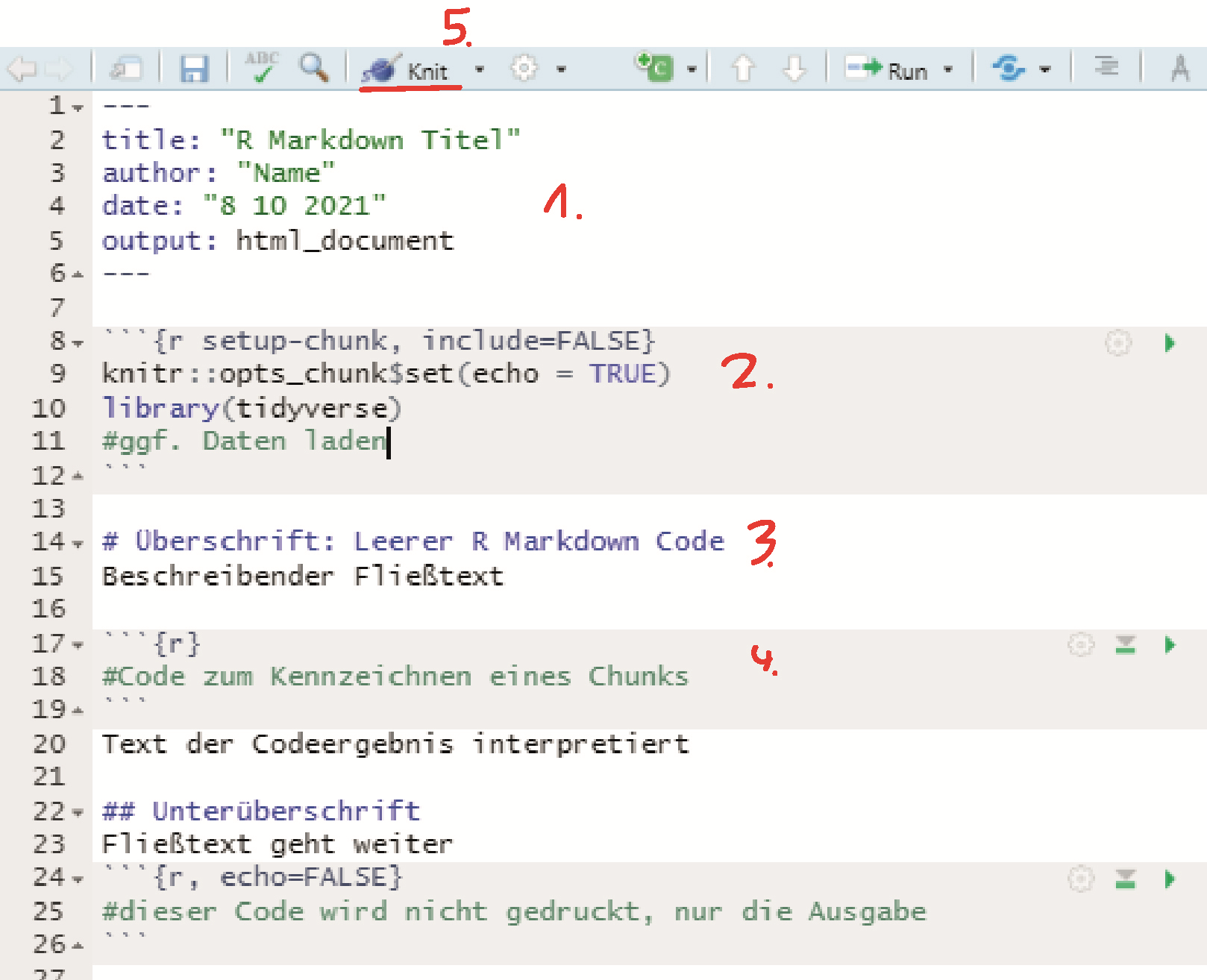
Knittet man dieses Skript mit dem Wollknäuel Button (5.) in der oberen Leiste, integriert es den ausgeführten Code mit dem Fließtext und druckt ein übersichtliches Dokument (html, pdf, txt oder doc). Das ist praktisch um z.B. Auswertungsergebnisse zu präsentieren.
- Im Header werden Titel und Dokumententyp für das Ausgabe-Dokument festgelegt
- Die Code Blöcke (
Chunks) sind mit je drei rückwärts gestellten Hochkommata (Backticks) am Anfang und Ende des Chunks eingerahmt. Werden sie vom R Markdown Skript als solche erkannt, wird auch die Hintergrundfarbe automatisch abgeändert. Im ersten Chunk solltenglobale Chunk Optionen festgelegt(z.B. Working Directory setzen oder ändern), alle notwendigenPackages geladenund dieDaten eingelesenwerden. - Den Fließtext kann man mit Überschriften (
#) und Unterüberschriften (##) strukturieren, im Code kennzeichnet#Kommentare - Zu Beginn eines Chunks muss man innerhalb einer geschwungenen Klammer spezifizieren(```{…}):
Es ist möglich Codechunks von anderen Programmiersprachen (z.B. Python oder TeX) einzubetten, Standard ist
r(optional) Nach einem Leerzeichen: Einzigartiger Chunk-Name
(optional) Nach einem Komma: Befehle, um die Ausgabe des Chunks in das neue Dokument zu steuern:
include = FALSEWeder Code noch Ergebnis erscheinenecho = FALSENur das Code-Ergebnis erscheintmessage = FALSENachrichten zum Code erscheinen nichtwarning = FALSEWarnungenzum Code erscheinen nichtfig.cap = "..."Hiermit lassen sich Grafiken beschriften
4.2 Hilfe
Sie merken, dass die Befehle und Funktionen zum Teil sehr spezifisch sind und Sie sich kaum alles behalten können. Am wichtigsten ist die Reihenfolge und Vollständigkeit der Zeichen: Vergessen Sie ein Komma, ein Backtick oder eine Klammer zu setzen, dann kann R den Code schon nicht interpretieren. Zum Glück erkennt R Studio das oft und weist einen darauf während des Codens mit einem roten x neben der Zeilennummer hin. Andernfalls erscheint eine Fehlermeldung beim Ausführen des Codes.
Wenn Sie den Namen einer Funktion oder eines Packages nicht direkt erinnern, können Sie den Anfang des Namens im Chunk oder in der Console eingeben, RStudio bietet einem nach einem kurzen Moment eine Liste möglicher Optionen an, aus der Sie wählen können. Haben Sie eine Funktion gewählt, können Sie die Tab-Taste drücken und es werden die verschiedenen Funktionsargumente angezeigt, um die Funktion zu spezifizieren, was oft sehr hilfreich ist. Möchten Sie wissen, was eine Funktion macht oder in welcher Reihenfolge die Funktionsargumente eingegeben werden, können Sie ?FUN in die Console eintippen, wobei FUN Platzhalter für den Funktionsnamen ist. Alternativ können Sie im Help-tab unten rechts suchen. Die Dokumentation ist oft sehr ausführlich.
Im Zweifelsfall haben Sie immer die Möglichkeit einschlägige Suchmaschinen im Internet zu verwenden. Oft werden Sie dabei auf StackOverflow weitergeleitet. Auf Englisch gestellte Fragen oder Probleme führen zu besseren Treffern. Noch trivialer ist es, im Skript des Kurses oder im eigenen Code nachzuschauen, die Tasten STRG + F habe ich schon 1000 Mal gedrückt, sie könnten dabei hilfreich sein. Falls Sie bei anderen Autoren nachlesen möchten, gibt es Bücher zu R, die meist sogar kostenlos online zur Verfügung stehen und einem eine Einführung in R geben: Z.B. R Cookbook oder R for Data Science.
4.3 Wiederholung: Werte & Vektoren
Datenformate in R sind von einfach zu komplex: Value, Vektor, matrix, (array), data.frame, tibble und list. Ihnen noch unbekannte Datenformate werde im Laufe des Kapitels erklärt.
Die kleinste Objekteinheit in R ist ein Value. Die unterschiedlichen Datentypen von Values sind bereits bekannt:
- Text, bzw. Charakter (
chr), auch String genannt, - numerische Werte (
num), auchdouble - logische Werte (
logi) - fehlende Werte (
NA),Not Available
Sie weisen einem Objektnamen einen Wert per <- zu (Shortkey: ALT + -), der Datentyp des Values wird automatisch erkannt.
Mit der Funktion mode() können Sie sich den Datentypen anzeigen lassen.
Vektoren reihen Werte desselben Datentyps auf c(Wert1, Wert2, ...):
# c() kombiniert die Werte zu einem Vektor, der dem Variablennamen vec1 zugewiesen wird:
vec1 <- c(3, 6, 3.4)Wenn Sie versuchen Elemenete mit unterschiedlichen Datentypen ein einen Vektor zusammenzufassen, z.B. c("kreativ", 3.5), wobei "kreativ" einen komplexeren Typ (Text ofer character) als 3.5 (numerisch oder double) besitzt, werden die Typen der Elemente vereinheitlich. Dabei wird der Typ des weniger komplexen Element in den Typ des komplexeren Elements umgewandelt, sodass alle Elemente des Vektors den komplexeren Datentyp besitzen. Die Datentypen-Übersichtstabelle ist von komplex zu einfach (1–4) geordnet. Die Umwandlung des Datentypen nennt sich coercion.
## [1] "kreativ" "3.5"3.5 wird in "" ausgegeben, der numerische Wert wurde in Text umgewandelt.
4.3.1 Coercion (Umwandlung von Typen)
Sie können den Datentypen auch per Funktion ändern, z.B. as.character(), as.double():
## [1] "1" "TRUE" "abc" "4.1627"## Warning: NAs introduced by coercion## [1] 2.0000 NA NA 4.1627Coercion gibt es auch in Matrizen, Arrays (Mehrdimensionale Matrizen) und in Spaltenvektoren von Datensätzen (data.frames und tibbles). Nur Listen können verschiedene Datentypen und Elemente enthalten list(Element1, Element2, ...). Das können Strings, Vektoren, aber auch Datensätze, Funktionen und sogar andere Listen sein.
4.3.2 Aufruf per Index von Daten aus mehreren Ebenen
Ihnen ist bekannt, dass Sie zum Zugriff auf einzelne Elemente deren Indexnummer verwenden können:
## [1] 3Das geht auch in verschachtelten Listen:
# Definiere eine Liste, die eine Liste und einen Vektor enthält:
# (Anm.: hier habe ich keine Namen für die Elemente vergeben)
mylist <- list(list(1, "a"),
vec_4)
# Rufe Element 1 der äußeren Liste: (1, "a"), und davon dann Element 2 ab:
mylist[[1]][2]## [[1]]
## [1] "a"Ich habe jetzt mehrere Variablen (Values, Vektoren, Listen) definiert, sie sind in meinem RStudio im Environment-tab oben rechts aufgetaucht.
4.3.3 Nullwerte
In (fast) jedem Experiment und jeder Erhebung kommt es mal vor, das Daten fehlen. Es kann sein, dass Versuchspersonen einen Fragebogen nicht ausgefüllt haben oder man die Schrift einfach nicht lesen konnte. Solche Werte müssen ebenfalls kodiert werden, aber entsprechend als fehlend. Wir haben bereits gesehen, dass wir solche fehlenden Werte in R mit dem Wert NA (“not availible”) kodieren können. R hat allerdings noch zwei weitere Arten von Nullwerten.
Neben NA gibt es auch noch NULL. Dieser Wert wird verwendet, wenn wir eine Variable definieren möchten, ihr aber noch keinen Wert zuweisen wollen. In diesem Fall können wir der Variable eine Art Platzhalter als Wert zuweisen, der gar keinen Wert hat, also quasi leer ist. Dieser “leere Wert” ist NULL.
Gleichzeitig können wir NULL verwenden, wenn wir ein Objekt aus einer list oder eine Spalte aus einem data.frame entfernen wollen – wir weisen dem Element einfach den Wert NULL zu:
Experiment_Reaktionszeit <- data.frame("x" = 1:10, "y" = 11:20)
Experiment_Reaktionszeit$x <- NULL
Experiment_Reaktionszeit## y
## 1 11
## 2 12
## 3 13
## 4 14
## 5 15
## 6 16
## 7 17
## 8 18
## 9 19
## 10 20Der letzte Nullwert, den R kennt, ist NaN (“Not a Number”). NaN taucht immer dann auf, wenn wir Zahlen nicht darstellen können oder das Ergebnis einer Berechnung nicht definiert ist, beispielsweise wenn wir versuchen, durch Null zu teilen oder den Logarithmus einer negativen Zahl berechnen. NaN ist technisch gesehen ein numerischer Wert – wir können ihn daher auch zusammen mit anderen numerischen Werten, beispielsweise in einem numerischen Vektor, verwenden.
4.4 Der Workspace
Rechts oben im Fenster ist das Environment-tab. Hier sieht man alle im global Workspace definierten Objekte (Datenstrukturen: Werte, Vektoren, Matrizen, Arrays, Listen, data.frames, tibbles; und Funktionen) aufgelistet:
Bei Werten, Vektoren und Matrizen steht sogar der Datentyp des Objektes mit dabei. Per Doppelklick können Sie die Objekte jeweils einzeln oben links im extra Fenster (Datenansicht-tab ) anschauen. rm(Objektname) ist die Funktion zum Entfernen einzelner Objekte aus dem globalen Workspace. Das Besensymbol im Environment-tab oben rechts fegt den globalen Workspace leer. Es ist zu beachten, dass R Markdown beim knitten nicht auf den globalen Workspace zugreift, sondern einen eigenen Workspace aus dem Code in den Chunks erstellt. Beim Ausführen einzelner Chunks per Markieren und STRG/CTRL&Enter oder grüner Pfeil rechts wird jedoch auf den globalen Workspace zugegriffen. Beim Schließen von RStudio werden Sie gefragt, ob Sie den globalen Workspace in die .RData als img speichern lassen, dann stehen die Objekte in der nächsten Sitzung wieder zur Verfügung, solange Sie dieselbe Projektdatei öffnen. Offene Skipte und offene Datenansicht-tabs werden beim Schließen ebenfalls mit der Projektdatei assoziiert. Geladene Packages gehen leider verloren, diese müssen Sie jedes Mal beim Starten von RStudio neu laden: library(Packagename). Deshalb ist es Konvention am Anfang jedes Skriptes erstmal die Packages zu laden. Haben Sie Objekte im Workspace gespeichert, können Sie deren Namen verwenden, um sich auf diese zu beziehen und z.B. weitere Berechnungen vorzunehmen.
4.5 Wiederholung: Einfache Berechnungen
x <- 5 # definiert den Wert der Variable x
y <- 5 # definiert den Wert der Variable y
x + y # Summe von x und y
x * y # Produkt von x und y
sqrt(x) # Wurzel aus x
x**(1/2) # x hoch 0.5
# Weise der Variable z das Ergebnis der Gleichung `x + y` zu. `z` erscheint im Workspace:
z <- x + y
# Multipliziere die Elemente von vec_4 mit 5 und speichere als Variable e:
e <- vec_4 * 5In R gibt es einige integrierte Funktionen um mit Vektoren zu rechnen:
4.5.1 Übersicht Berechnungsfunktionen
Folgende Funktionen können Sie auf num-Vektoren und Matrizen anwenden, je nach Funktion auch auf chr Vektoren oder Datensätze, wobei diese sich dann meist nur auf die Einträge in der oberen Ebene, z.B. auf die Anzahl der Spalten und nicht auf die Spalteneinträge beziehen.
| Funktion | Bedeutung | Funktion | Bedeutung |
|---|---|---|---|
min(x) |
Minimum | mean(x) |
Mittelwert |
max(x) |
Maximum | median(x) |
Median |
range(x) |
Range | var(x) |
Varianz |
sort(x) |
sortiert x |
sd(x) |
Standardabweichung |
sum(x) |
Summe aller Elemente | quantile(x) |
Quantile von x |
cor(x, y) |
Korrelation
von x und y |
length() |
Länge von x |
4.5.1.1 Beispiel einer z-Standardisierung eines Vektors mit 3 Einträgen
# Def. der Variable geschwister:
geschwister <- c(8, 4, 12)
# MW:
mw_geschwister <- mean(geschwister)
mw_geschwister## [1] 8## [1] 4# z-Standardisierung des Vektors:
z_geschwister <- (geschwister-mw_geschwister) / sd_geschwister
z_geschwister## [1] 0 -1 14.6 Matrizen
Matrizen sind 2D-Datenstrukturen, sie bestehen aus Vektoren gleicher Länge und enthalten einen Datentyp. Mit dem
Befehl matrix() können sie erstellt werden:
# Erstellt die Matrix bsp_mat mit 4 Zeilen, 4 Spalten und leeren Einträgen:
bsp_mat <- matrix(NaN, nrow = 4, ncol = 4) # NaN (Not a Number) ist vom Typ `num`
bsp_mat## [,1] [,2] [,3] [,4]
## [1,] NaN NaN NaN NaN
## [2,] NaN NaN NaN NaN
## [3,] NaN NaN NaN NaN
## [4,] NaN NaN NaN NaNIch habe eine 4×4 Matrix erstellt, die mit NaNs gefüllt ist. Hätte ich diverse Datentypen zugeordnet, wären diese zum komplexeren coerced worden. Auf Werte in Matrizen kann mit matrixname[Zeilennummer, Spaltenummer] zugegriffen werden. Praktischerweise stehen die entspechenden Indizes neben der oben angezeigten Matrix. Beispiele zum Auswählen und Zuweisen neuer Werte:
# Weil Spalte 1 von bsp_mat und vec_4 dieselbe Länge haben,
# kann ich der Spalte 1 die Einträge von vec_4 zuweisen.
# Dadurch, dass der Eintrag für die Zeilennummer leer ist,
# beziehe ich mich auf alle Zeilen:
bsp_mat[ , 1] <- vec_4
bsp_mat## [,1] [,2] [,3] [,4]
## [1,] 1 NaN NaN NaN
## [2,] 3 NaN NaN NaN
## [3,] 3 NaN NaN NaN
## [4,] 7 NaN NaN NaN# Recycling: Wird einem Bereich ein einzelner Wert zugeordnet,
# wird dieser vervielfacht (wie oben bei NaN):
bsp_mat[ , 2] <- 8
bsp_mat## [,1] [,2] [,3] [,4]
## [1,] 1 8 NaN NaN
## [2,] 3 8 NaN NaN
## [3,] 3 8 NaN NaN
## [4,] 7 8 NaN NaN## [,1] [,2] [,3] [,4]
## [1,] 1 8 0 NaN
## [2,] 3 8 1 NaN
## [3,] 3 8 0 NaN
## [4,] 7 8 1 NaNCoercion: TRUE wurde zu 1 und FALSE wurde zu 0. Wenn man nun eine bestimmte Zeile oder Spalte betrachten möchte, kann man dies auch über die Indizierung tun, hierbei kann man sich beliebig austoben. Die Regeln dafür sind dieselben wie bei Vektoren, nur in 2D, wobei stets [Zeile, Spalte] gilt.
## [1] 1 3 3 7## [1] 7Hier wird es turbulent:
## [,1] [,2] [,3] [,4]
## [1,] 1 8 0 NaN
## [2,] 3 8 0 NaN## [,1] [,2] [,3]
## [1,] 8 1 NaN
## [2,] 8 0 NaN
## [3,] 8 1 NaNDa ich jetzt Bereiche der Matrix auswählen kann, kann ich vielleicht Berechnungen vornehmen:
## [1] "numeric"## [,1] [,2] [,3] [,4]
## [1,] 1 8 0 NaN
## [2,] 3 8 1 NaN
## [3,] 3 8 0 NaN
## [4,] 7 8 1 NaN## [1] 0 5 0 1Es sind immer noch nicht angegebene Nummernwerte in der Matrix. Da ich mich beim Berechnen auf Bereiche der Matrix beschränke, die vergebene numerische Werte haben (Spalten 1 bis 3 ohne NaN Einträge), habe ich ein sinnvolles Ergebnis bekommen. Was passiert, wenn ich mit NaN Einträgen rechnen möchte?
## [1] 1 8 0 NaN## [1] NaNDie Summe der Zeile führt zu keinem interpretierbaren Ergebnis. Zum Auslassen der NaNs wird das Funktionsargument na.rm = TRUE verwendet (rm steht für remove):
## [1] 9## [1] 4Nun, da wir mit dem Rechnen in Matrizen vertraut sind, möchte ich die letzte Spalte mit Einträgen füllen:
# Speichere bsp_mat unter sav_mat zur späteren Verwendung:
sav_mat <- bsp_mat
# Weise Spalte 4 einen `chr`-Vektor zu:
bsp_mat[,4] <- c("coercion", "kann", "nervig", "sein")
bsp_mat## [,1] [,2] [,3] [,4]
## [1,] "1" "8" "0" "coercion"
## [2,] "3" "8" "1" "kann"
## [3,] "3" "8" "0" "nervig"
## [4,] "7" "8" "1" "sein"## [1] "character"Konnte ich eben noch den Mittelwert einer Spalte bilden, so geht das jetzt nicht mehr, da alle Einträge der Matrix zu chr coerced wurden. In einem typischen Datensatz sind aber Variablen verschiedener Typen (num und chr) enthalten. Dieses Problem ließe sich mit Listen lösen, welche aber unübersichtlich sind. Datensätze bestehen manchmal aus unüberschaubar vielen Einträgen und deshalb sollten sie wenigstens übersichtlich strukturiert sein.
4.7 tidy Daten
Es gibt eine Konvention dafür, wie man Datensätze, die mehreren Beobachtungseinheiten (Fällen) verschiedene Parameter (Variablen) zuordnet. Wichtig für die eigene strukturierte Arbeit ist in erster Linie Konsistenz, z.B. dass Sie bei Variablennamen aus mehreren Wörtern immer den Unterstrich als Trennzeichen verwenden. Es hat sich als überlegen für die Auswertung von Daten herausgestellt, Fälle in Zeilen und Variablen in Spalten einzuordnen. Dieses Prinzip dürfte einigen schon von SPSS bekannt sein.
| Variable1 | Variable2 | Was ist ‘tidy’ data? | |
|---|---|---|---|
| Fall1 | Wert11 | Wert12 | Eine Zeile pro Beobachtung |
| Fall2 | Wert21 | Wert22 | Eine Spalte pro Variable |
| Fall3 | Wert31 | Wert32 | Eine Tabelle pro Untersuchung |
| Fall4 | Wert41 | Wert42 | eindeutige Namen |
| Fall5 | Wert51 | Wert52 | Konsistenz |
| Fall6 | Wert61 | Wert62 | … |
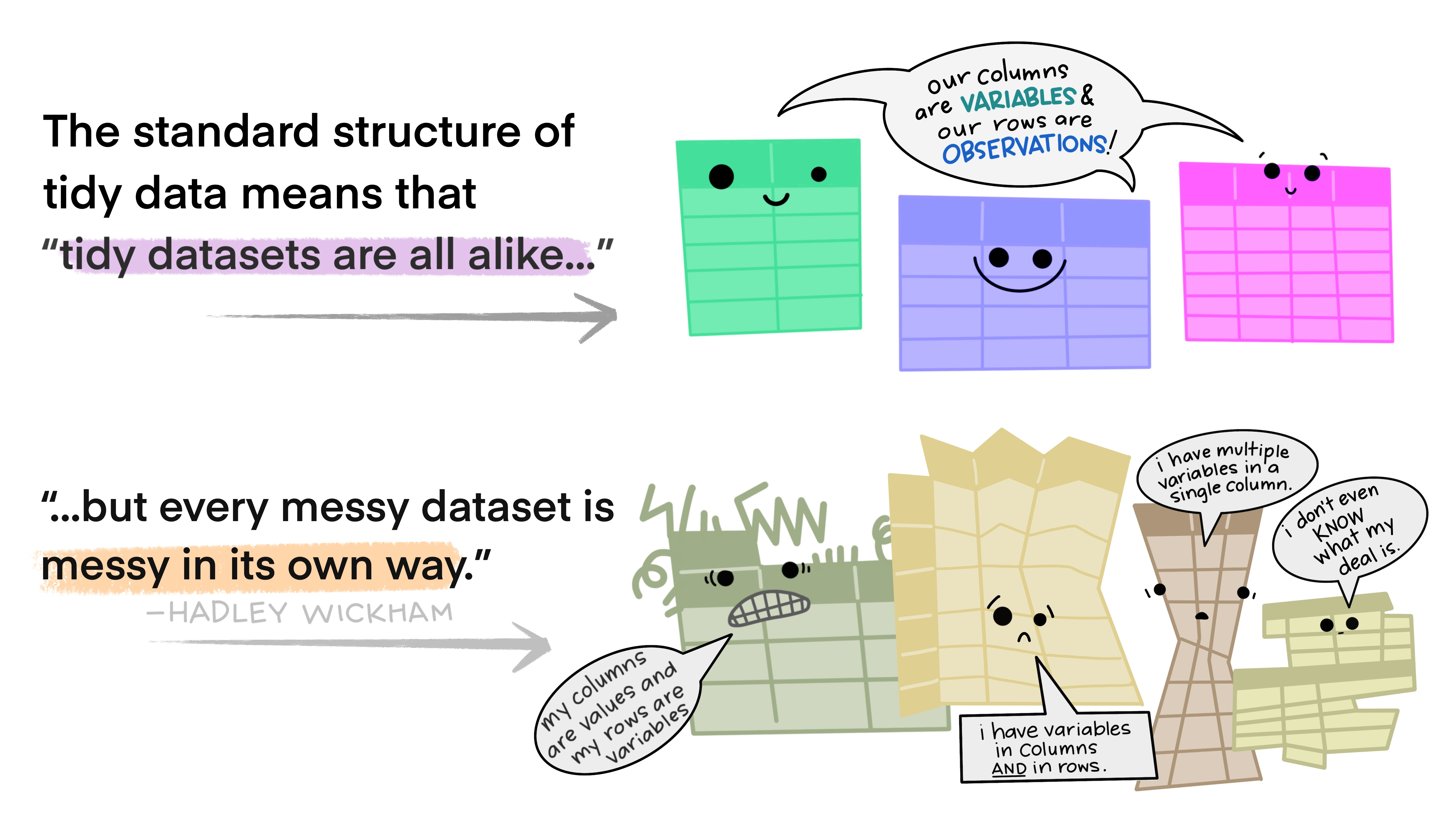
Illustration by Allison Horst
Es gibt noch weitere Koventionen und Empfehlungen für konsistentes und ordentliches Arbeiten in R und mit Datensätzen im Allgemeinen, z.B. dass man keine Farbcodierungen verwenden sollte. Vorerst genügt es, wenn Sie sich an die Basics hier halten. Diese Art Daten zu strukturieren lässt sich im data.frame und noch besser im tibble umsetzen: Beides sind Tabellen mit Spaltenvektoren, die jeweils verschiedene Datentypen enthalten können. Deswegen stellen beide das bevorzugte und für unsere Zwecke wichtigste Datenformat dar.
4.7.1 tidyverse
Bevor wir uns dem übersichtlichsten Datenformat, den tibbles widmen, müssen wir das entsprechende Package einmalig in der Console installieren. Ich habe den Code auskommentiert, weil das Package bei mir bereits installiert ist:
# R kennt den Namen von zu installierenden Packages noch nicht, deswegen in "":
#install.packages("tidyverse")Das Package tidyverse enthält mehrere nützliche Packages, die eine saubere Datenverarbeitung zum Ziel haben. Packages müssen bei jeder Sitzung neu aktiviert bzw. angehängt werden. Für Sie relevante Packages im tidyverse sind tibble, readr, stringr, dplyr, purr und ggplot2.
4.8 data.frames (df) und tibbles (tib)
Beides sind Tabellen mit Spaltenvektoren (Variablen), die je verschiedene Datentypen enthalten können. Hier zunächst die Übersicht über die Funktionen zum Managen des Datensatzes:
| Funktion zum | data.frame() |
tibble() |
|---|---|---|
| Datenformat konvertieren | as.data.frame() |
as_tibble() |
| Definieren | data.frame(var1, ...) |
tibble(var1, ...) |
| Aufrufen des Datensatzes | df |
tib |
| Auswählen einer Variable | df$var |
tib$var |
| Auswählen eines Bereiches | df[rowIdx, colIdx] |
tib[rowIdx, colIdx] |
| Definieren neuer Variablen | df$var_neu <- c(...) |
tib$var_neu <- c(...) |
| Ergänzen von Zeilen | rbind(df, Zeilen) |
rbind(tib, Zeilen) |
| Ergänzen von Spalten | cbind(df, Spalten) |
cbind(tib, Spalten) |
| Zeilennamen vergeben | row.names(df) <- c("name1", ...) |
relocate(tib, namevec) |
Sie können die beiden Datensatz-Formate einfach in das jeweils andere konvertieren. Datensätze lassen sich auch per Formel definieren: data.frame() oder tibble(), wobei hier die Spaltenvektoren aneinandergereiht werden. Es bietet sich an, dabei direkt Namen für die Spaltenvektoren zu vergeben:
# Erzeuge einen data.frame durch Verwendung der Spaltenvektoren aus vorherigen Matrizen,
# denen Namen zugewiesen werden (Denken Sie beim Definieren der Spaltenvektoren daran
# diese mit einem Komma voneinander zu trennen):
test_df <- data.frame("text" = bsp_mat[ , 4],
"ist_Verb" = sav_mat[ , 3])
test_df## text ist_Verb
## 1 coercion 0
## 2 kann 1
## 3 nervig 0
## 4 sein 1In Bezug auf weitere Funktionen des Packages tidyverse sind tibbles ein wenig praktischer. Große tibbles werden beim Aufrufen etwas übersichtlicher angezeigt (nur die ersten 10 Zeilen).
## # A tibble: 4 × 2
## text ist_Verb
## <chr> <dbl>
## 1 coercion 0
## 2 kann 1
## 3 nervig 0
## 4 sein 1Einzelne Spalten können ganz einfach aufgerufen werden, in dem man den $-Operator benutzt. Schreibt man diesen direkt hinter den Namen des Datensatzes, klappt automatisch eine Liste mit allen Spalten auf:
## [1] "coercion" "kann" "nervig" "sein"Es ist auch möglich, mehrere Zeilen und/oder Spalten auszugeben. Dies funktioniert wie bei Matrizen per Indexnummer:
## # A tibble: 3 × 1
## text
## <chr>
## 1 kann
## 2 nervig
## 3 seinDie Adressierung einzelner Spalten und Zeilen ermöglicht dann zum Beispiel die Berechnung von Kennwerten nur für einzelne Spalten. Z.B. kann man die Kosten für Konzertkarten im Jahr 2022 aufsummieren lassen:
# Definiere ein tibble mit 2 Variablen:
tickets_2022 <- tibble("Artist" = c("Ed Sheeran", "Billy Ellish", "The Weeknd", "Dua Lipa",
"Imagine Dragons"),
"Kosten" = c(79.32, 282, 116, 136, 68.71))
# Berechne die Summe einer dieser beiden Variablen:
sum(tickets_2022$Kosten)## [1] 682.03Der $-Operator wird für fast alle höheren Datentypen verwendet, um auf diese zuzugreifen. Dies gilt zum Beispiel auch für die meisten Outputs von Funktionen (t-Test, Anova, SEMs) und Listen, es müssen aber wie im tibble, Namen für die Listeneinträge vergeben worden sein:
# Erstelle eine Liste mit diversen Objekten aus meinem Workspace und vergebe Namen
# (über welche Sie später auch auf die Listeneinträge zugreifen können):
list_of_thingis <- list(tibbi = test_tib,
ticki = tickets_2022,
geschwi = geschwister,
vari = var1)
# Per $-Operator und Name in der Liste wird ein Eintrag gewählt:
list_of_thingis$geschwi## [1] 8 4 12## NULL## [1] "Ed Sheeran" "Billy Ellish" "The Weeknd"
## [4] "Dua Lipa" "Imagine Dragons"Mir fällt auf, dass ich den Namen einer Künstlerin in tickets_2022 falsch geschrieben habe, das möchte ich ändern:
# $-Operator und Indexing per Nummer lassen sich auch kombinieren:
tickets_2022$Artist[2] <- "Billy Eilish"Sie können also nicht nur Elemente aus Datensätzen abrufen, sondern diese mit dem <- neu zuweisen. Sie können das $ auch verwenden um ganz neue Spalten in die Datensätze einzufügen:
# Definiere eine neue Spalte im Datensatz:
tickets_2022$Priorität <- c(2, 4, 3, 5, 1)
# Besser ae statt ä im Variablennamen:
tickets_2022$Prioritaet <- tickets_2022$Priorität
tickets_2022## # A tibble: 5 × 4
## Artist Kosten Priorität Prioritaet
## <chr> <dbl> <dbl> <dbl>
## 1 Ed Sheeran 79.3 2 2
## 2 Billy Eilish 282 4 4
## 3 The Weeknd 116 3 3
## 4 Dua Lipa 136 5 5
## 5 Imagine Dragons 68.7 1 1Nun gibt es eine Spalte zu viel. Ich möchte sie wieder löschen - vorsichtig hiermit(!):
Mit den Funktionen rbind() und cbind() lassen sich die data.frames und tibbles um gleichbreite bzw. gleichlange Vektoren oder Datensätze ergänzen. Verbinden Sie ein tibble und einen data.frame miteinander, werden diese zum Format tibble coerced. Zunächst füge ich zwei weitere Zeilen hinzu, dann zwei weitere Spalten:
# Zwei weitere Konzerte in neunem data.frame:
extra_Konzerte <- data.frame("Artist" = c("Elton John", "Coldplay", "RHCP"),
"Kosten" = c(139.00, 259.00, 200),
"Prioritaet" = c(2.7, 2.3, 1.7))
# Zeilenweises Aneinanderbinden:
tickets_2022 <- rbind(tickets_2022, extra_Konzerte)
# Zwischenergebnis:
tickets_2022## # A tibble: 8 × 3
## Artist Kosten Prioritaet
## <chr> <dbl> <dbl>
## 1 Ed Sheeran 79.3 2
## 2 Billy Eilish 282 4
## 3 The Weeknd 116 3
## 4 Dua Lipa 136 5
## 5 Imagine Dragons 68.7 1
## 6 Elton John 139 2.7
## 7 Coldplay 259 2.3
## 8 RHCP 200 1.7# Ort und Begleitung in neuem tibble:
ort_begleitung <- tibble("Ort" = c("Frankfurt", "London", "Muenchen", "Berlin", "Gdynia", "Frankfurt", "Frankfurt", "Koeln"),
"Begleitung" = c("Juergen", "Samu", "Anni", "Jan und Laura", "Oma", "Papa", "Schwester", "die Girls"))
# Spaltenweises Aneinanderbinden:
tickets_2022 <- cbind(tickets_2022, ort_begleitung)
# Endergebnis:
tickets_2022## Artist Kosten Prioritaet Ort Begleitung
## 1 Ed Sheeran 79.32 2.0 Frankfurt Juergen
## 2 Billy Eilish 282.00 4.0 London Samu
## 3 The Weeknd 116.00 3.0 Muenchen Anni
## 4 Dua Lipa 136.00 5.0 Berlin Jan und Laura
## 5 Imagine Dragons 68.71 1.0 Gdynia Oma
## 6 Elton John 139.00 2.7 Frankfurt Papa
## 7 Coldplay 259.00 2.3 Frankfurt Schwester
## 8 RHCP 200.00 1.7 Koeln die GirlsEin Unterschied zwischen tibbles und data.frames ist, dass tibbles keine Zeilennamen kennen. Das vereinfacht das Format. Möchten Sie trotzdem gerne Zeilennamen vergeben, müssen Sie sich mit einer neuen Variable (z.B. namevec) behelfen, die Sie mit relocate(tib, namevec) an den Anfang des Datensatzes stellen können.
# Definiere ein einfaches tibble mit Zeilennamen in einer Spalte:
newtib <- tibble("sp1" = c(1, 2, 3), "namevec" = c("Zeile1", "Zeile2", "Zeile3"))
newtib## # A tibble: 3 × 2
## sp1 namevec
## <dbl> <chr>
## 1 1 Zeile1
## 2 2 Zeile2
## 3 3 Zeile3## # A tibble: 3 × 2
## namevec sp1
## <chr> <dbl>
## 1 Zeile1 1
## 2 Zeile2 2
## 3 Zeile3 3Mit dem Hinzufügen und Abändern und Vertauschen von Spaltenvektoren haben wir schon ein bisschen an der Oberfläche der Möglichkeiten zur Datenaufbereitung gekratzt, die in der nächsten Sitzung ausführlich behandelt werden. Jetzt, wo Sie mit dem Management von Datensätzen vertraut sind, wollen wir vorhandene Datensätze einlesen:
4.9 Einlesen und Speichern von Daten
Daten können in R Studio auf unterschiedliche Weise eingelesen werden.

Es gibt Packages mit frei verfügbaren Datensätzen, z.B. einen Datensatz zu Pinguinen: palmerpenguins.
Horst AM, Hill AP, Gorman KB (2020). palmerpenguins: Palmer Archipelago (Antarctica) penguin data. R package version 0.1.0. https://allisonhorst.github.io/palmerpenguins/
Nach einmaliger Installation des Packages (install.packages("palmerpenguins") muss es geladen werden:
# Jedes Mal beim Durchlaufen des Skripts soll das Package angehängt werden
# (ohne "", weil R das Package bereits installiert hat und kennt):
library(palmerpenguins)
# penguins ist zwar schon ein tibble, aber im Workspace rechts ist es nicht zu sehen,
# daher weise ich es dem Namen pingus zu:
pingus <- penguinsIn der Regel werden Sie aber einen selbst erhobenen oder einen aus dem Internet heruntergeladenen Datensatz einlesen wollen. Mein Tipp ist, den Datensatz in das Working Directory zu speichern, dann finden Sie ihn schneller und er ist in der Nähe Ihrer Auswertung. Noch eleganter ist es einen Unterordner namens data in den Ordner des Working Directories anzulegen, in den Sie alle Datensätze zu ihrem Projekt speichern können. Im File-tab unten rechts navigieren Sie zu der Datei mit dem Datensatz und dann klicken Sie diese zum Importieren des Datensatzes an (alternativ können Sie im Environment-tab über den Button Import Dataset einen Datensatz zum Importieren auf ihrem Computer suchen). In RStudio erscheint ein Fenster zum Importieren, unten rechts wird der automatisch der dem Dateiformat und unten links angegebenen Optionen entspricht, ggf. werden sogar benötigte Packages geladen.
Um einen Datensatz per Code zu importieren sind Dateiformat, die Trennzeichen (sep) und die Dezimalzeichen (dec) besonders relevant. Das Standard-Dateiformat ist .csv, hier sind Kommata Trennzeichen (sep=",") und Punkte kennzeichnen Dezimalstellen (dec="."). Sie können die Funktionen read_cvs() oder read_delim() für dieses Dateiformat verwenden, letztere sollte Trenn- und Dezimalzeichen automatisch erkennen. Hier ist eine Übersicht zu den Einlesefunktionen in base R (also ohne zusätzlich geladene Packages) und im tidyverse Package, der Unterschied ist, dass base R Funktionen die Daten in einen data.frame laden, tidyverse Funktionen in ein tibble:
| Funktion zum | sep |
dec |
in base R | im tidverse |
|---|---|---|---|---|
| autolesen | auto | "." |
read.delim() |
read_delim() |
| autolesen | auto | "," |
read.delim2() |
read_delim2() |
| lesen von | "," |
"." |
read.csv() |
read_csv() |
| lesen von | leer | "." |
read.table() |
read_table() |
| schreiben | "," |
"." |
write.csv() |
write_csv() |
Wichtigstes und oft einziges Funktionsargument ist der vollständige Dateiname, er wird in " angegeben. Alternativ können Sie statt dem Dateinamen auch die Funktion file.choose() angeben, die dann ein interaktives Fenster zur Dateiauswahl öffnet. Falls Sie die Datei in einem Unterordner vom Working Directory gespeichert haben (ich erstelle mir meist einen Unterordner namens data), wird der Name des Unterordners mit einem / dem Dateinamen vorangestellt. Vergessen Sie nicht, innerhalb der Anführungszeichen sowohl den Namen des Datensatzes als auch das Dateiformat nach einem Punkt zu nennen (z.B.“data/Datensatz1.csv”). Das Einlesen von Daten funktioniert nur, wenn der einzulesende Datensatz per <- einem Namen zugewiesen wird. Beispiel zum Laden eines .csv Datensatzes:
# Lese meinen socken.csv Datensatz aus dem Unterordner data in ein tibble namens socken:
socken <- read_delim("data/socken.csv")
socken ## # A tibble: 2 × 3
## Stoff Gewicht Bewertung
## <chr> <dbl> <dbl>
## 1 Seide 0.03 10
## 2 Wolle 0.08 9Excel Dateien werden mit Funktionen read_excel(), read_xls() oder read_xlsx() aus dem Package readxl, SPSS Dateien mit der Funktion read_svs() aus dem Package haven eingelesen. Auch zum Einlesen von SAS, Stata oder anderen Dateiformaten gibt es entsprechende Funktionen. Die Standardfunktion zum Abspeichern von Datensätzen in eine Datei ist write_csv(), bzw. in base R write.csv(), da dieses Dateiformat die beste Kompatibilität mit anderer Software aufweist. Beim Speichern müssen Sie neben dem Dateinamen und ggf. dem Dateipfad noch den Namen des Datensatzes, den Sie speichern möchten, als erstes Funktionsargument angeben:
Es gibt noch ein weiteres erwähnenswertes Dateiformat, das von R selbst: .RDS. Die Funktionen saveRDS() und readRDS() bieten die beste Funktionalität in R.
4.10 Datensätze (dat) anschauen
Um sich einen geladenen Datensatz komplett anzuschauen, können Sie diesen im Workspace anklicken, oder deren Namen an die Funktion view(dat) übergeben. Der Datensatz pingus hat 344 Zeilen, das kann ich im Workspace sehen. Da er als tibble gespeichert ist, könnte ich diesen per Name aufrufen (nur die ersten 10 Zeilen würden dargestellt werden). Mit der Funktion head(dat) wird einem der Kopf des Datensatzes ausgegeben, genau genommen die ersten 6 Zeilen. Die Funktion ist besonders nützlich für große data.frames, da diese beim Aufrufen per Name die Console überfüllen.
## # A tibble: 6 × 8
## species island bill_…¹ bill_…² flipp…³ body_…⁴ sex year
## <fct> <fct> <dbl> <dbl> <int> <int> <fct> <int>
## 1 Adelie Torge… 39.1 18.7 181 3750 male 2007
## 2 Adelie Torge… 39.5 17.4 186 3800 fema… 2007
## 3 Adelie Torge… 40.3 18 195 3250 fema… 2007
## 4 Adelie Torge… NA NA NA NA <NA> 2007
## 5 Adelie Torge… 36.7 19.3 193 3450 fema… 2007
## 6 Adelie Torge… 39.3 20.6 190 3650 male 2007
## # … with abbreviated variable names ¹bill_length_mm,
## # ²bill_depth_mm, ³flipper_length_mm, ⁴body_mass_gEinen Überlick über die Datenstruktur, inklusive Factor-levels (der Faktorstufen) erhalten Sie mit der Funktion str(dat):
## tibble [344 × 8] (S3: tbl_df/tbl/data.frame)
## $ species : Factor w/ 3 levels "Adelie","Chinstrap",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ island : Factor w/ 3 levels "Biscoe","Dream",..: 3 3 3 3 3 3 3 3 3 3 ...
## $ bill_length_mm : num [1:344] 39.1 39.5 40.3 NA 36.7 39.3 38.9 39.2 34.1 42 ...
## $ bill_depth_mm : num [1:344] 18.7 17.4 18 NA 19.3 20.6 17.8 19.6 18.1 20.2 ...
## $ flipper_length_mm: int [1:344] 181 186 195 NA 193 190 181 195 193 190 ...
## $ body_mass_g : int [1:344] 3750 3800 3250 NA 3450 3650 3625 4675 3475 4250 ...
## $ sex : Factor w/ 2 levels "female","male": 2 1 1 NA 1 2 1 2 NA NA ...
## $ year : int [1:344] 2007 2007 2007 2007 2007 2007 2007 2007 2007 2007 ...Zeile 1 gibt Auskunft über Größe und die Klasse des Objektes, tibbles sind eine Art data.frame. In den weiteren Zeilen werden die Datentypen bzw. Faktorlevel und die ersten Werte der Spaltenvektoren angezeigt.
4.10.1 Häufigkeit von Factorlevels
Faktoren und die Zuweisung mit der Funktion factor() kennen Sie bereits aus dem vorherigen Kapitel. Die Funktion levels(Faktor) gibt die möglichen Ausprägungen einer Faktorvariable wieder. Faktoren eignen sich oft besser als Vektoren zum Plotten, Gruppieren oder für Häufigkeitstabellen. Mit der Funktion count(dat, var) lassen sich beispielsweise die Häufigkeiten der Levels eines Faktors ausgeben:
## # A tibble: 3 × 2
## island n
## <fct> <int>
## 1 Biscoe 168
## 2 Dream 124
## 3 Torgersen 52Mit der ersten deskriptiven Statistik zu einem Datensatz in R schließen wir dieses Kapitel ab. Es folgen geschickte Methoden zur Datenaufbereitung.